新闻中心
News center
nature子刊|基于知识图确定EGFR突变型非小细胞肺癌耐药的驱动因素
来源:
|
作者:星
|
发布时间: 652天前
|
1143 次浏览
|
分享到:
今天要为大家介绍的是一篇基于知识图的推荐框架确定EGFR突变型非小细胞肺癌耐药的驱动因素的高分SCI文章,发表在nature computational science。本文新颖之处在于文中证明了一个基于多目标优化方法的推荐系统可以用于在二次耐药的情况下重新排序CRISPR,帮助将基因命中的优先排序时间从几个月减少到几分钟。大大提高筛选效率!
本案例适合
1.人工智能(AI),计算机科学(CS),大数据(DS)
2.跨专业学习算法的同学,专业为生物信息工程,生命科学,医学统计学,医学
科研星级
数 据:⭐ ⭐ ⭐ ⭐ ⭐
方 法:⭐ ⭐ ⭐ ⭐
应用度:⭐ ⭐ ⭐ ⭐
今天要为大家介绍的是一篇基于知识图的推荐框架确定EGFR突变型非小细胞肺癌耐药的驱动因素的高分SCI文章,发表在nature computational science。本文新颖之处在于文中证明了一个基于多目标优化方法的推荐系统可以用于在二次耐药的情况下重新排序CRISPR,帮助将基因命中的优先排序时间从几个月减少到几分钟。大大提高筛选效率!

对EGFR抑制剂(EGFRi)的耐药是治疗非小细胞肺癌(NSCLC)的主要障碍。寻找潜在耐药性标记的有效方法是进行功能性基因筛选,如CRISPR。这种从CRISPR筛选中筛选出“高价值”点击的筛选过程涉及手工筛选,为更快地找到耐药性的关键驱动因素,整合临床前、临床和文献证据的异构生物医学知识图谱上建立了一个推荐系统。推荐系统根据基因与EGFRi耐药的潜在机制联系起来,对基因进行排名。这种无偏的方法从> 3000个基因中识别出57个抗性标记,将命中时间从几个月缩短到几分钟。
概述
这项研究探索了如何将一个生物学问题转化为一个推荐系统问题。为说明这一点,在本研究中重点研究了肺癌的耐药问题。目标是建立一个推荐的解决方案,找到驱动耐药性的关键基因。
本研究关注携带表皮生长因子受体(EGFR)激活突变的非小细胞肺癌(NSCLC)。使用第一代或第二代EGFR酪氨酸激酶抑制剂治疗初期肿瘤患者迅速产生耐药性。实验设置模拟了诊所的治疗场景,耐药细胞的生长或阳性选择被测量并用于定义驱动耐药的机制。一旦耐药性建立,这些细胞就可以成为潜在的目标。
除纯粹的生物特征,我们的推荐系统依赖于一个专门构建的异构生物医学知识图谱的数据。知识图为实体(节点)之间的关系(边)提供了方便的概念表示。通过将图源特征与临床特征相结合,可以发现导致肺癌耐药的不明显基因。
文中证明了一个基于多目标优化方法的推荐系统可以用于在二次耐药的情况下重新排序CRISPR,帮助将基因命中的优先排序时间从几个月减少到几分钟。
方法
EGFRI CRISPR屏幕设计
对EGFR突变细胞系PC-9和HCC827以及EGFR T790M二次耐药突变的吉非替尼抗性克隆进行全基因组CRISPR敲除和激活筛选。将靶向18010个人类基因的sgRNA文库转导到细胞系。选择文库转导细胞后,分别用吉非替尼或奥希替尼处理21天,以筛选抗性基因。
crispr聚合屏幕分析
采用fastqc76和muttiqc77评价测序数据的质量。结果的原始计数是利用基尼系数控制每个样本的阅读深度、样本内引导RNA的多样性以及样本的预期聚类。
CRISPR Screen QC和必要性评估
作为我们CRISPR筛选处理框架的一部分,我们开发了一个执行最终QC检查的管道,以确保筛选具有良好的质量,并与我们功能性基因组学中心执行的其他筛选相媲美。该分析评估了是否可以从屏幕中预测出预期的必要基因。
CRISPR screen-derived特性
共确定了一个>3000抗性基因的起始列表,作为重新排序的命中点。总结一致性度量标准被定义为细胞系的总数量,其中一个基因出现了一次成功。hit是根据可取性得分来定义的。我们使用两种不同类型的阈值来定义CRISPRn和CRISPRa屏幕的命中次数,分别考虑了CRISPRn和CRISPRa屏幕的一致性,以及屏幕的整体一致性。
Graph-derived特性
利用RESCAL算法在全图的基础上进行嵌入计算。全图还用于计算描述性网络指标,如节点度和连接到一个节点的唯一邻居的数量。文中进一步关注了蛋白质-蛋白质相互作用(PPI)亚图。
完美主义特征
为评估一个基因参与EGFR抑制剂耐药性的总体文献支持,分析了PubMed和PMC文章的语料库,确定了两个关键术语——“EGFR”和“NSCLC”。还计算了在先前定义的(癌症和耐药性)背景下的多术语基因共发生。这个分析背后的想法是发现在EGFR抑制剂耐药性环境中倾向于共同发生的基因集。利用基因共现矩阵在SkywalkR app中构建交互式热图。
结果
CRISPR点击率的重新排序可以作为多目标优化
将CRISPR点击率的重新排序构建为一个多目标优化问题。形式目标是同时优化k个目标,体现在k客观的功能:fi (x), f2 (x)……(x)。单个函数形成一个矢量函数F(x):
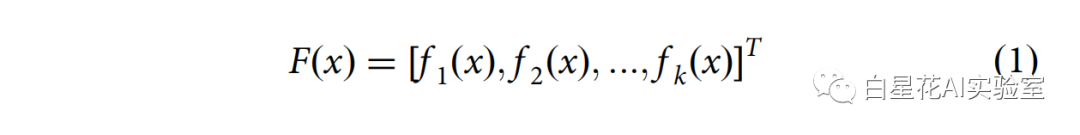
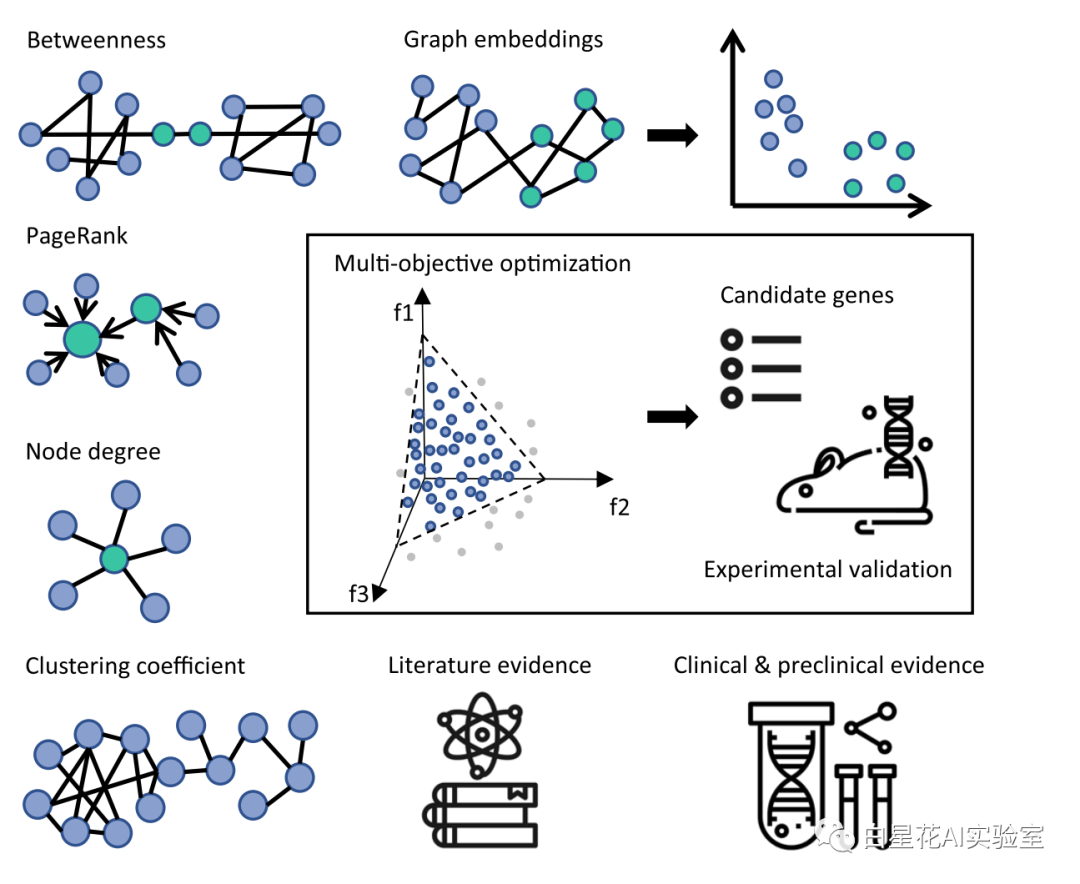
式中x = [x1, X2,…], Xm] EΩ;X表示决策变量,Ω-decision变量空间。因此,多目标优化可定义为目标函数集F(x)的最小化(或最大化)。通过计算帕累托前沿多样化的组基于CRISPR筛选数据和其他支持证据定义的目标,可以缩小EGFR TKI耐药标志物的数量(图1)
图(1)
一组混合功能支持推荐系统
为支持推荐系统,文中组装了一组混合的丰富特征(图1),每个特征代表一个目标。所选择的特征与NSCLC中EGFR抑制剂耐药性相关,并与不同的证据相对应。以下为关键特征类型以及考虑它们对CRISPR点击率重新排序的理由。
CRISPR:CRISPR屏幕数据是重新排名的起点。本研究依赖类似EGFR突变肺癌临床治疗场景的筛选,使用患者群体中常见的具有EGFR突变的NSCLC癌细胞系,这些癌细胞系使用第1代或第3代EGFR抑制剂治疗。共有7个基于一致性的特征被纳入特征集:(1)3个基于屏幕失去功能部分的特征;(2)基于屏幕的函数增益部分的三特征;(3)全屏反映总体一致性的汇总度量
Graph-derived特性:本研究使用自定义知识图(KG)作为推荐系统的信息来源。KG包含1100万个节点和8400万个边,由37个公共和内部数据集组成,例如Hetionet, OpenTargets, ChEMBL和Ensembl32。通常,知识图捕获的生物实体之间的交互模式可以转化为特征并被推荐系统使用一个多种方式(图1)。
当一个目标被优先用于药物开发时,需要确保:(1)基因原则上是可处理的;(2)一个基因不应该是必需的
综上所述,最终的混合集包含27个丰富的特征,混合特征空间的相关分析表明:(1)结构图特征之间存在较强的正相关关系;(2)敲除和激活屏衍生的CRISPR特征呈负相关。
交互界面允许专家重新排序CRISPR点击:在现实场景中,对于同一变量的特定目标集和优化方向的选择因专家而异。为适应不同观点,并使领域科学家能够在探索目标之间权衡,文中构建了一个交互式应用程序——SkywalkR (图2)47SkywalkR应用程序结合了不同方面的知识,以指导CRISPR的优先级重新排序,以进行实验验证。它激发对可能性的探索,突出现有知识的差距,并激励调整对最优解决方案的预期。通过SkywalkR交互界面实现的自动化工程的丰富功能与多目标优化大大减少了时间所需基因优先级从几周到几分钟。
图(2)

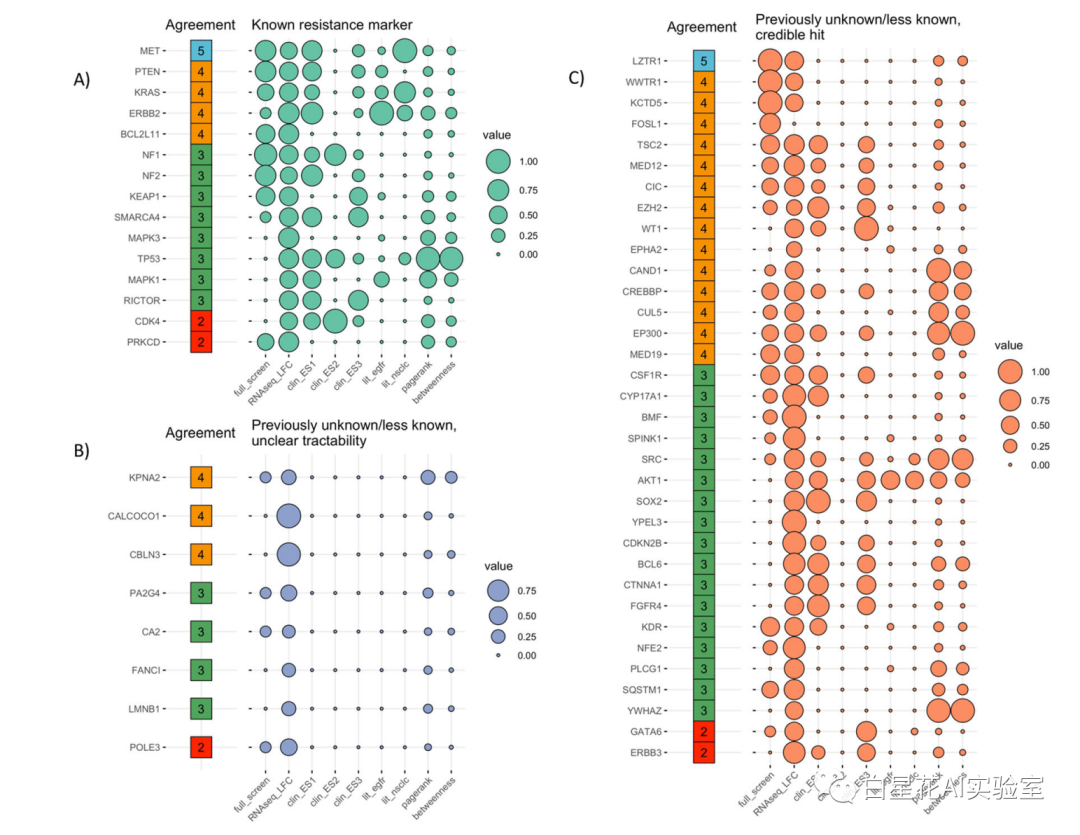
为根据专家意见评估推荐框架,文中修正了一组默认的首选项。选择默认值集是为了模拟领域专家CRISPR hit验证的过程,也包含从文献中提取的图衍生特征和汇总指标,得到的列表包含57个推荐基因(图3)。
文中为每个基因分配了最常见的标签,产生了三个主要类别:“已知”、“先前未知、可信命中”和“先前未知命中、不清晰的可追踪性”。分析了与默认首选项中包含的目标对应的值。为便于比较,将值进行了标准化(图3)。
图(3)
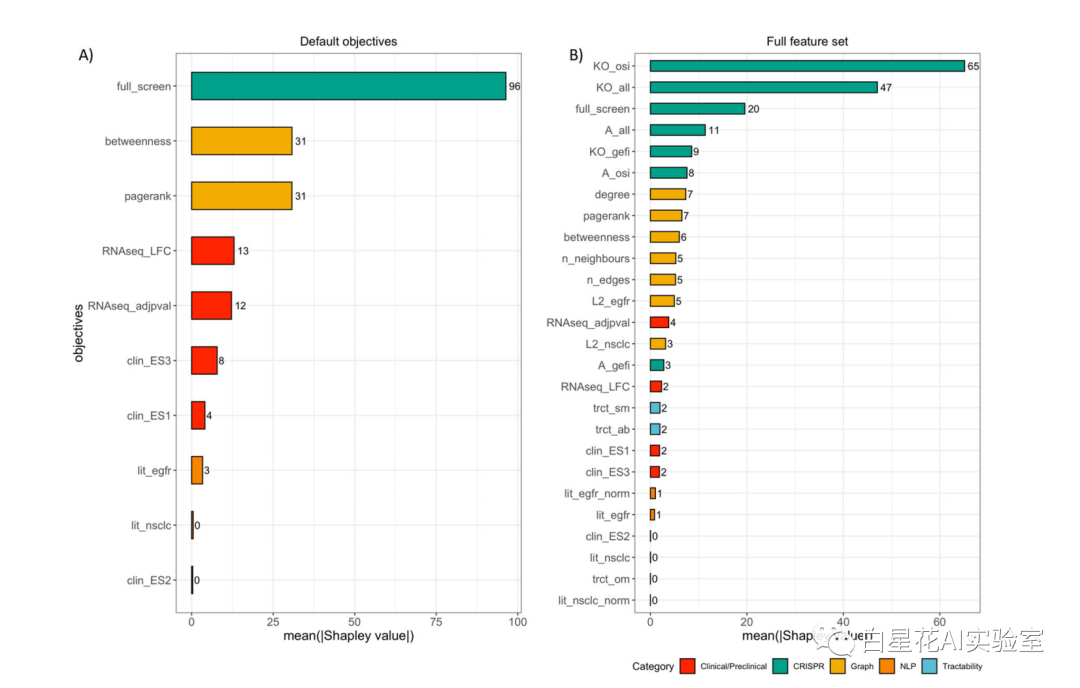
Shapley表明crispr衍生特征的影响很大
为分配正面标签,使用了一组排序为二级CRISPR screen38的100个基因,并训练了两个随机森林模型:
(1) 基于默认特征子集;
(2) 基于包括临床、临床前、文献、CRISPR和图形衍生类别在内的全套丰富特征(图4)
总的来说在两个实验中,CRISPRderived特征对基因分类的影响最大。
图(4)
网络分析和临床知识表明,推荐的基因中有EGFR耐药机制
为了将优先排序的命中位点与已知的EGFR生物学联系起来,我们进行了途径富集分析。证实了“肺癌对EGFR抑制剂的耐药机制”在顶级富集通路中。
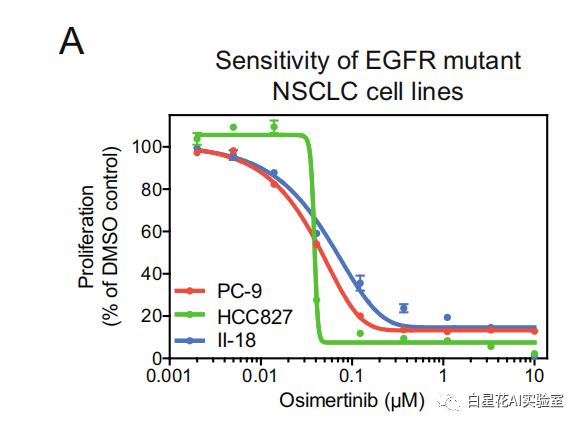
实验验证了表观遗传和ras信号基因在介导抗性表型中的关键调控作用
为进一步验证推荐基因的一个子集,通过实验研究了它们对奥西替尼耐药性的直接影响。为了提供基本事实,将已建立的EGFRi耐药标志物作为验证研究(MET和PTEN)的背景。对6个推荐基因(MET、WWTR1、EZH2、PTEN、NF1和KCTD5)在EGFR突变NSCLC细胞系中对奥希tinib敏感(图5A)。
图(5)A
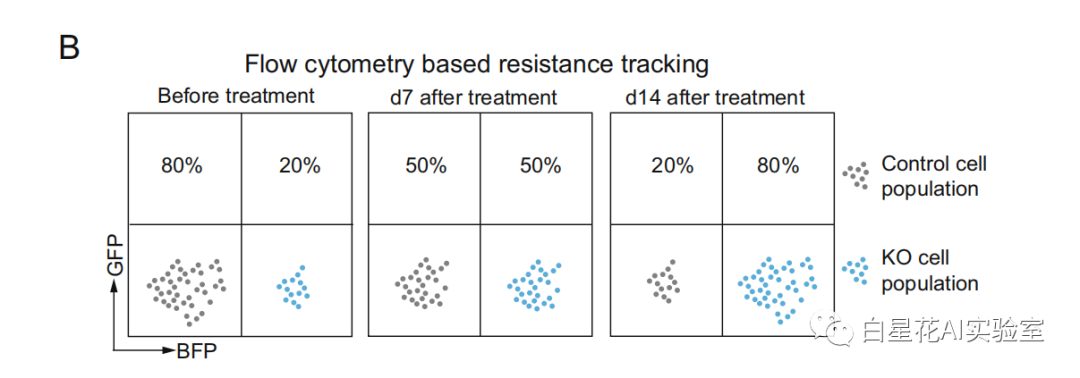
PTEN, NF1和KCTD5先前被证明负调控MAPK或PI3K/AKT信号53-55,这是EGFR TKI耐药的已知驱动因素。因此预期PTEN、NF1和KCTD5的调控可能介导稳定的耐药表型。来验证假设一个基于流式细胞术的长期竞争分析(图5B)。
PTEN, NF1和KCTD5先前被证明负调控MAPK或PI3K/AKT信号53-55,这是EGFR TKI耐药的已知驱动因素。因此预期PTEN、NF1和KCTD5的调控可能介导稳定的耐药表型。来验证假设一个基于流式细胞术的长期竞争分析(图5B)。
图(5)B
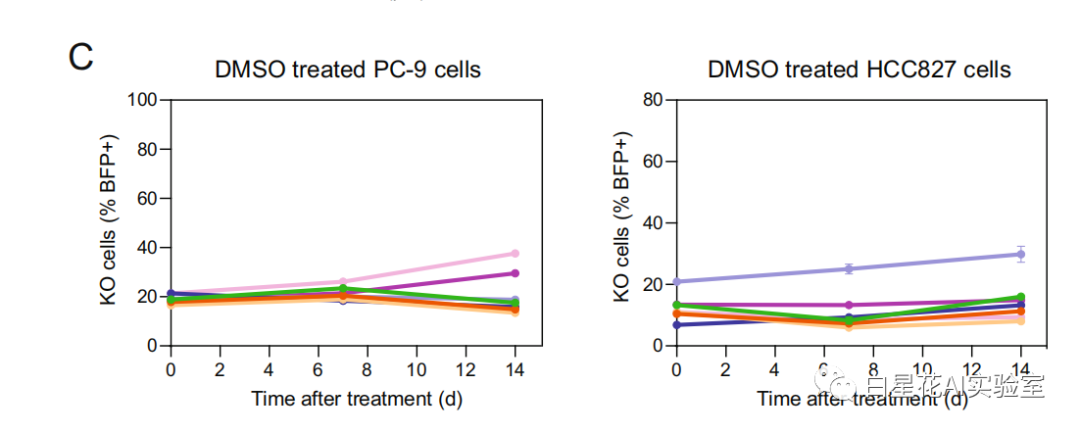
实验表明,在对照(DMSO)条件下共培养14天后,与非靶向对照(NTC)细胞相比,干扰NF1、PTEN或KCTD5的表达(图5B, C)不影响增殖。
图(5)C
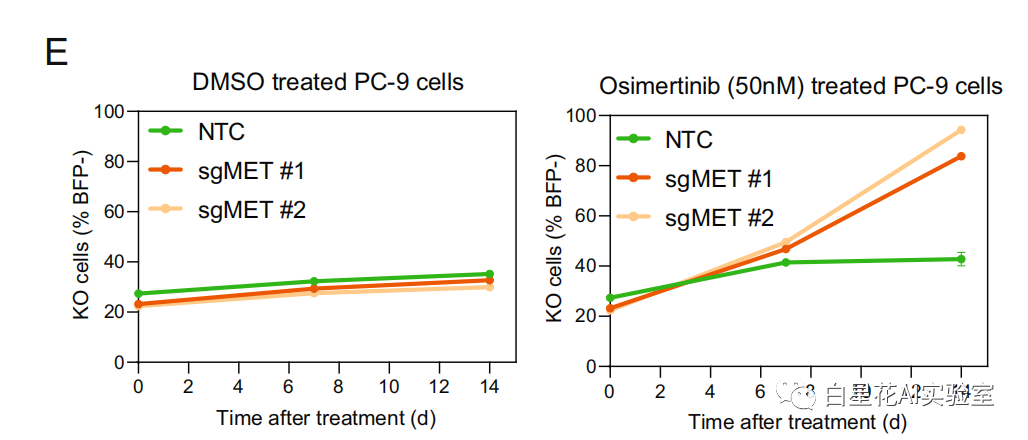
正如预期的那样,在控制条件下,过表达MET不会改变细胞增殖。相比之下,与对照修饰细胞相比,奥希替尼处理下的细胞增殖显著升高(图5E)。
图(5)E
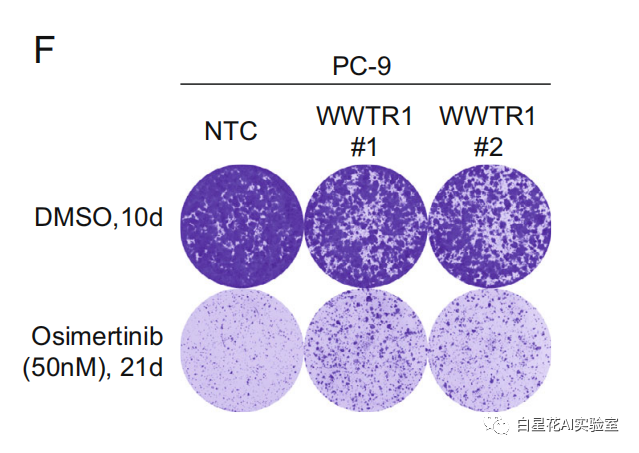
长期克隆实验表明,与NTC对照细胞相比,PC-9中WWTR1的激活(图5F)导致了对奥希替尼治疗具有耐药性的菌群的大量生长。
图(5)F
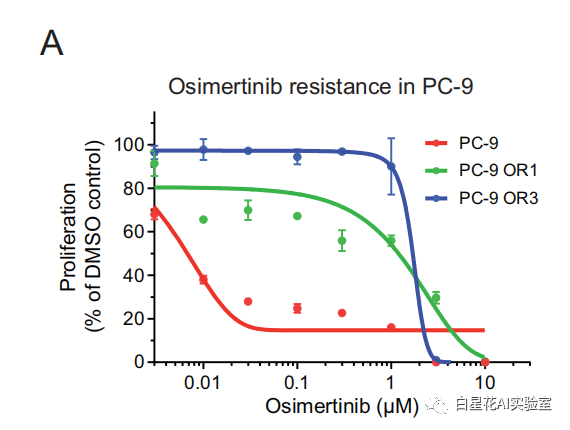
为了验证SRC作为电阻驱动的有效性,使用抗性细胞系(图6A),绘制了三种小分子SRC抑制剂的剂量-反应曲线,比较了亲本克隆和抗性克隆的敏感性。
图(6)A
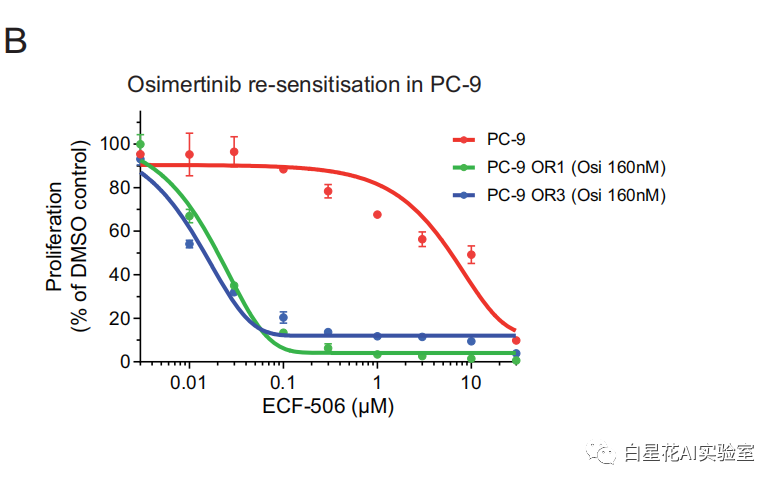
亲代细胞系一般对SRC抑制剂治疗的单一治疗效果具有耐药性(图6B)。使用等量的这些SRC抑制剂治疗可以使所有对奥希替尼耐药(OR)的PC-9、HCC827、NCI-H1975和HCC4006细胞系对奥希替尼临床相关剂量(160 nM)敏感,从而突出SRC在介导奥希替尼耐药中的重要性。
图(6)B
增加浓度的奥希替尼与EZH2抑制剂他美司他联合使用显示对奥希替尼的耐药性增加呈剂量依赖性。EZH2功能的丧失可以增加奥希替尼的耐药性(图6C)。
图(6)C
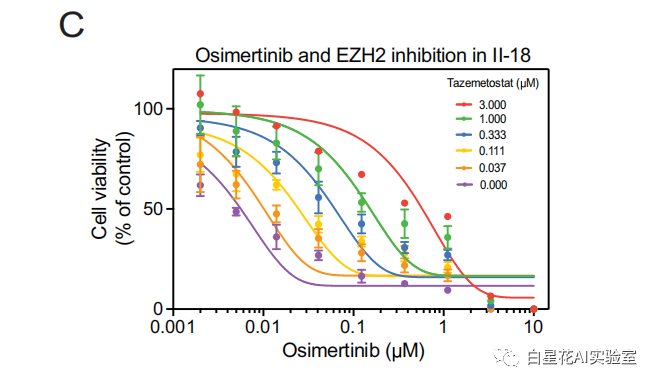
文中收集了初步的实验证据,表明操作推荐的ezh2、KCTD5、MET、NF1、PTEN、SRC和WWTR1基因亚群会导致奥希tinib耐药。这些观察结果表明,文中推荐的方法不仅可以发现众所周知的耐药性标记,而且还可以识别出以前未被探索和有希望的耐药性驱动因素。
讨论
大量生物医学数据的积累,以及对其理解和推理的需要,使得生物医学应用成为推荐技术有吸引力的领域。然而,将传统的推荐方法直接翻译到生物医学领域并非总是微不足道的。具体的问题空间和生物系统的复杂性需要有效的建议解决方案,可以在无监督或弱监督的设置中操作。文中认为更广泛地采用和系统地使用推荐系统来解决生物问题,具有改变生物医学研究和药物发现的潜力。

官方微信号
官方微信公众号

联系我们
合作单位
相关推荐
网站首页

Copyright 版权所有 © 白星花AI实验室 京ICP备2021038475号